Perché gli antibiotici funzionino come dovrebbero
I ricercatori del Fare all'ETH di Zurigo hanno decodificato la struttura della grande subunità del ribosoma mitocondriale fino al dettaglio atomico. Ciò fornisce una visione senza precedenti dell'architettura molecolare di questo ribosoma e della modalità d'azione degli antibiotici.
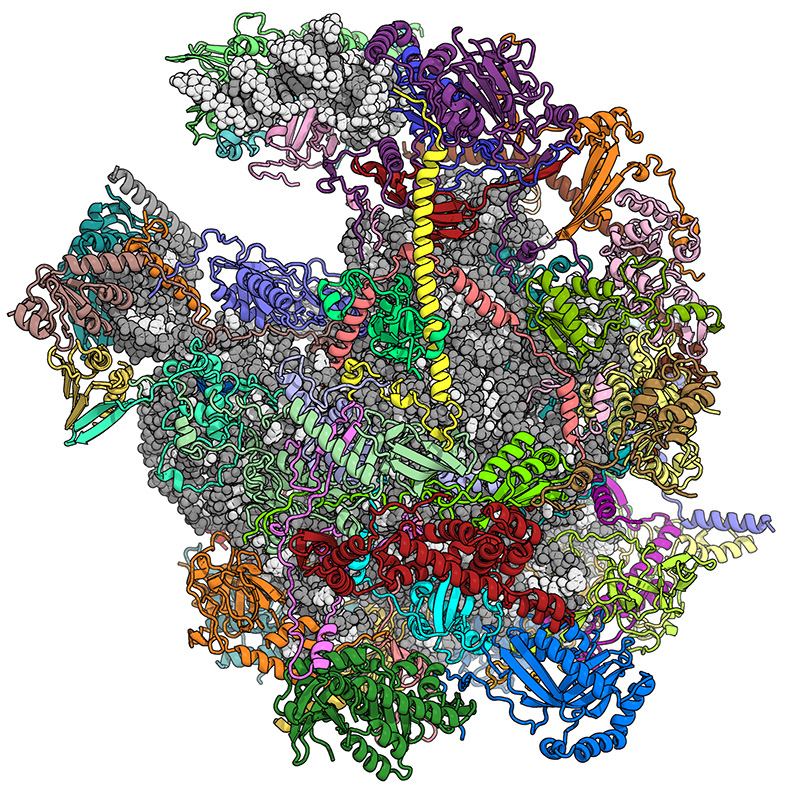
Un team di ricercatori dell'ETH di Zurigo, guidato dai professori Nenad Ban e Ruedi Aebersold, ha studiato all'ETH di Zurigo la struttura molecolare estremamente complessa dei mitoribosomi, i ribosomi dei mitocondri. I ribosomi sono presenti nelle cellule di tutti gli organismi viventi. Tuttavia, gli organismi superiori (eucarioti), che comprendono funghi, piante, animali e l'uomo, hanno ribosomi molto più complessi dei batteri. Negli eucarioti, i ribosomi possono essere divisi in due tipi: quelli nel citosol, la parte più consistente della cellula, e quelli nei mitocondri, le centrali elettriche della cellula. I mitocondri si trovano solo negli eucarioti.
I ribosomi fungono da dispositivi di decodifica e sono strettamente coinvolti nel processo di formazione delle proteine. Ogni ribosoma è composto da due subunità. La subunità più piccola utilizza gli acidi ribonucleici di trasferimento (RNA di trasferimento o tRNA) per decodificare il codice genetico, che viene fornito sotto forma di RNA messaggero. La subunità più grande assembla gli aminoacidi forniti dall'RNA di trasferimento come perline in una catena proteica.
Una risoluzione ancora più alta, ancora più dettagli
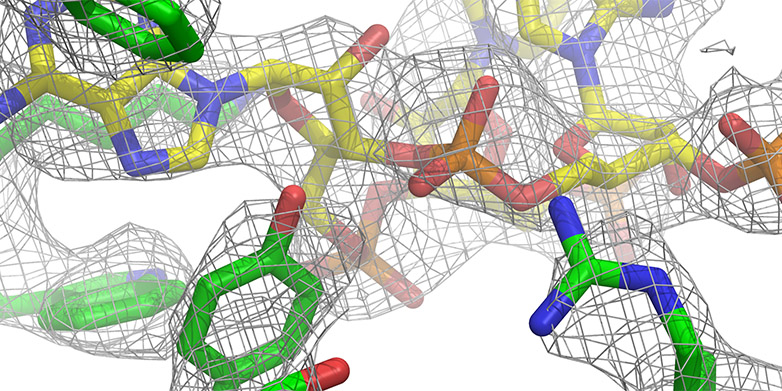
I ribosomi mitocondriali sono particolarmente difficili da studiare perché sono presenti solo in numero ridotto e sono difficili da isolare. All'inizio dell'anno, i ricercatori dell'ETH avevano già delucidato la struttura molecolare della subunità grande del mitoribosoma delle cellule di mammifero con una risoluzione di 4,9 angstrom (meno di 0,5 nanometri). Tuttavia, questa risoluzione non era abbastanza alta da permettere di umwelt und Geomatik un modello atomico affidabile della struttura allora sconosciuta. Il team del professor Nenad Ban dell'ETH è ora riuscito a farlo. Sono riusciti a decifrare l'intera struttura a 3,4 angstrom (0,34 nanometri). Gli scienziati hanno appena pubblicato i loro risultati sulla rivista scientifica "Nature".
Per le indagini sono stati utilizzati la microscopia crioelettronica ad alta risoluzione del Centro di microscopia elettronica dell'ETH di Zurigo (ScopeM) e metodi di spettrometria di massa all'avanguardia. Grazie ai recenti progressi tecnici nella microscopia crioelettronica e allo sviluppo di telecamere elettroniche in grado di correggere anche i minimi movimenti, è diventato possibile registrare biomolecole con una risoluzione inferiore a quattro angstrom.
Migliorare l'effetto degli antibiotici
Le nuove immagini mostrano in particolare la vista dettagliata del centro della peptidil transferasi (PTC), cioè il luogo in cui i blocchi di costruzione degli amminoacidi vengono uniti. Le proteine sintetizzate passano poi per un tunnel attraverso il quale lasciano infine la grande subunità del ribosoma.
"Questo processo è importante dal punto di vista medico", spiega Basil Greber, primo autore dello studio e dottorando del gruppo di Nenad Ban. Questo perché il tunnel viene utilizzato come punto di attacco per alcuni antibiotici: L'antibiotico si deposita nel tunnel e impedisce alle proteine appena prodotte di lasciarlo. Tuttavia, gli antibiotici sono progettati per inibire la sintesi proteica solo nei ribosomi dei batteri.
"Per poter essere utilizzato nell'uomo, un antibiotico non deve attaccare i ribosomi umani", spiega Greber. Gli antibiotici possono inibire la sintesi proteica solo nei ribosomi batterici. Il problema, tuttavia, è che i ribosomi mitocondriali sono simili a quelli dei batteri. Per questo motivo alcuni antibiotici interferiscono anche con i ribosomi mitocondriali. "Grazie alle scoperte dei ricercatori dell'ETH, in futuro sarà possibile progettare antibiotici che inibiscano solo i ribosomi batterici e non quelli mitocondriali. Questo è un prerequisito fondamentale per il loro utilizzo in clinica.
Scoperta sorprendente
I ricercatori dell'ETH hanno fatto anche una scoperta inaspettata. Hanno scoperto che i mitoribosomi utilizzano l'RNA di trasferimento in due modi in principio diversi. In primo luogo, utilizzano il tRNA per selezionare l'amminoacido giusto per la sintesi del peptide nella PTC. In secondo luogo, a differenza di tutti gli altri ribosomi, un tRNA è saldamente incorporato nella struttura. Questo sostituisce funzionalmente un acido ribonucleico (RNA) che è andato perso nel corso dell'evoluzione. Da tempo si sapeva che i ribosomi mitocondriali avevano integrato nuove proteine nella loro struttura durante lo sviluppo. Tuttavia, questa è la prima volta che si osserva una molecola di RNA completamente nuova. "Questo dimostra quanto i mitoribosomi siano adattabili", sottolinea Greber.
Nella sua ricerca, l'équipe dell'ETH affronta ora il grande compito, ancora irrisolto, di determinare la struttura della subunità più piccola del ribosoma mitocondriale. Poiché questa è più flessibile della subunità grande, si tratta di una sfida ancora più grande.
Letteratura di riferimento
Greber BJ, Boehringer D, Leibundgut M, Bieri P, Leitner A, Schmitz N, Aebersold R, Ban N: La struttura completa della subunità grande del ribosoma mitocondriale dei mammiferi. Nature, pubblicazione online dal 1° ottobre 2014. doi: pagina esterna10.1038/nature13895
Greber B et al: Architecture of the large subunit of the mammalian mitochondrial ribosome. Nature 2014, 505: 515-519. doi: pagina esterna10.1038/nature12890