Nous voyons actuellement les algorithmes informatiques révolutionner la biologie
L'intelligence artificielle permet de prédire la structure 3D des protéines. Bientôt, il devrait être possible de développer des protéines artificielles sur mesure grâce à de tels algorithmes, écrit Beat Christen.

Les algorithmes informatiques soutiennent la recherche biomédicale depuis des décennies et leur importance n'a cessé de croître. Mais ce à quoi nous assistons actuellement est un saut quantique qui éclipse tout ce que nous avons connu jusqu'à présent et qui aura des conséquences insoupçonnées : Grâce aux algorithmes de l'intelligence artificielle (IA), il est devenu possible de prédire, uniquement à partir de la séquence linéaire des éléments constitutifs des protéines - les acides aminés - quelle structure tridimensionnelle cette chaîne d'éléments constitutifs va former, et ce avec une extrême précision.
Pour en comprendre la signification, il faut savoir qu'en biologie, au niveau cellulaire, il s'agit en fait toujours de l'interaction spatiale des molécules, et cette interaction est déterminée par leur structure tridimensionnelle. Ce n'est que lorsque nous connaissons les structures et les interactions que nous pouvons comprendre la biologie. Et ce n'est que lorsque nous connaissons la structure des molécules que nous pouvons développer de manière ciblée des médicaments qui influencent la fonction de ces molécules.
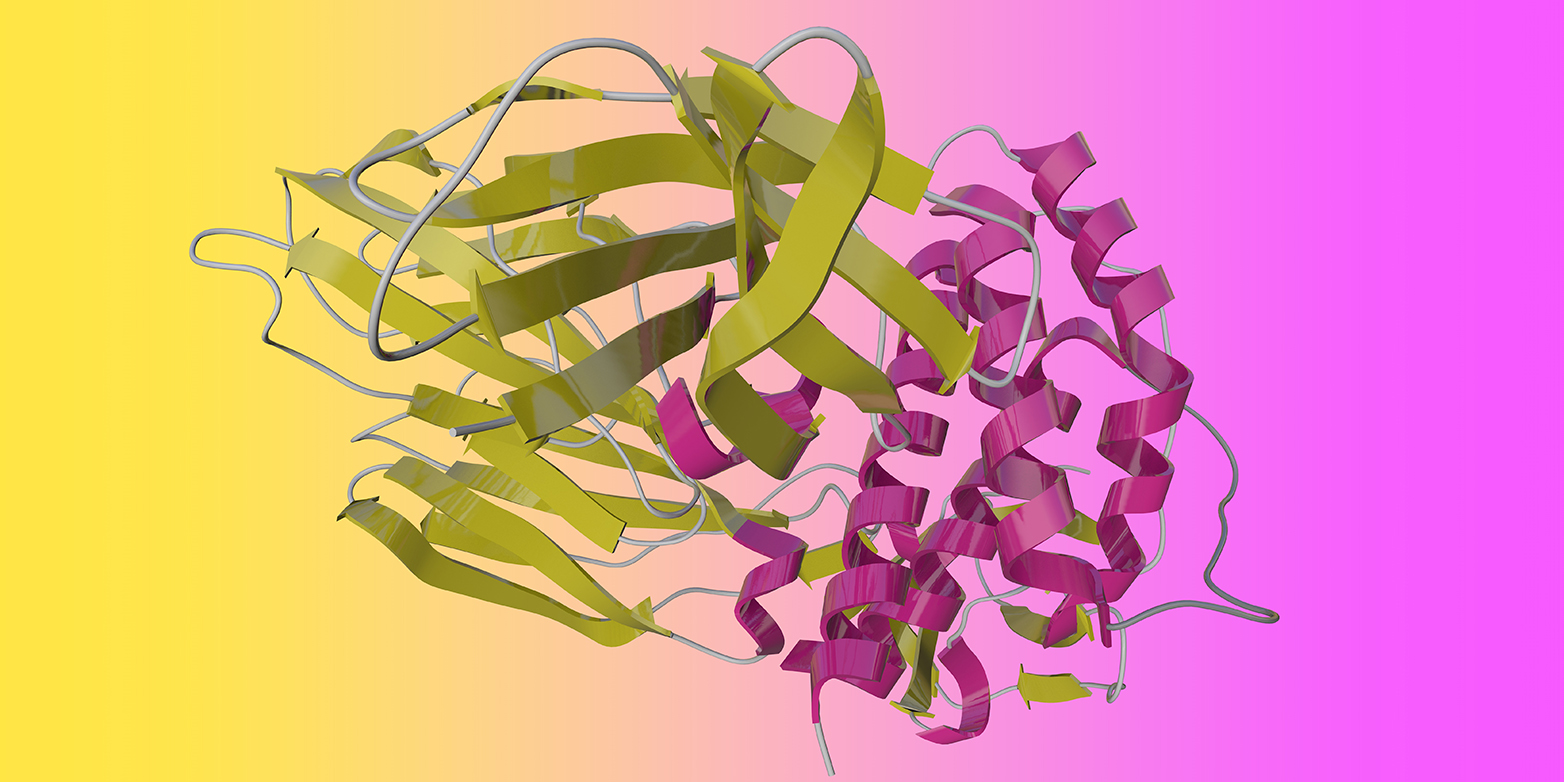
Jusqu'à présent, trois méthodes expérimentales permettaient de déterminer la structure tridimensionnelle des protéines : Analyse structurelle des rayons X, résonance magnétique nucléaire ainsi que, depuis quelques années, la cryo-microscopie électronique. L'arrivée d'une quatrième méthode précise avec l'IA n'est pas seulement liée à l'amélioration des algorithmes d'IA et aux grandes capacités de calcul disponibles aujourd'hui. Pour que l'IA puisse faire des prédictions précises, elle a également besoin de très nombreuses données d'excellente qualité avec lesquelles elle peut être entraînée. Ce saut quantique a donc été rendu possible grâce à d'immenses progrès et efforts dans les deux domaines : la science des données et la recherche expérimentale sur les protéines.
Concurrence de la recherche privée et publique
Actuellement, les projecteurs sont surtout braqués sur le programme d'IA Alphafold de Deepmind, une entreprise appartenant au groupe Google. Celle-ci est sans doute actuellement l'acteur le plus important dans le domaine de la prédiction de la structure des protéines. Dans le débat public, on oublie toutefois souvent que Deepmind est loin d'être le seul acteur dans ce domaine et que d'excellentes recherches sont menées notamment autour de David Baker de l'Université de Washington.
Cette concurrence entre recherche privée et publique a probablement stimulé et fait progresser le champ de recherche dans son ensemble. Bien entendu, les acteurs privés gardent pour eux nombre de leurs découvertes, par intérêt commercial. Néanmoins, la recherche hautement compétitive a également conduit à une immense amélioration des algorithmes d'IA publiés, que l'ensemble de la communauté scientifique peut désormais utiliser et développer. Je m'attends à ce que cette tendance se poursuive. Bientôt, les algorithmes d'IA nous fourniront des structures extrêmement précises pour toutes les protéines que nous connaissons. Cela nous permettra de concevoir d'autres médicaments ciblés sur ordinateur.
"La prochaine étape logique sera maintenant d'utiliser les algorithmes d'IA dans le sens inverse".Beat Christen
A partir d'une structure moléculaire tridimensionnelle développée sur ordinateur, il devrait être possible à l'avenir de calculer avec l'IA une séquence d'acides aminés qui s'assemblent exactement pour former la structure souhaitée avec la fonction moléculaire voulue.
Une fois cette séquence d'acides aminés déterminée, mon domaine de recherche entre en jeu. Je m'occupe du développement de gènes et de génomes artificiels et j'utilise également des algorithmes informatiques. En partant des séquences d'acides aminés, nous calculons la manière dont elles sont traduites en séquences d'éléments génétiques, c'est-à-dire en ADN. Et ce, de manière à ce que ces gènes puissent être facilement produits artificiellement dans la pratique.
Flux d'informations inversé
Il devrait donc bientôt être possible, à partir de n'importe quelle structure protéique tridimensionnelle développée sur ordinateur, de calculer un gène artificiel et de le synthétiser. Du point de vue biotechnologique, il sera ainsi possible de produire des protéines artificielles dans des micro-organismes, par exemple de nouvelles substances pharmaceutiques actives, des vaccins ou des enzymes intéressantes pour l'industrie.
Depuis les tout premiers êtres vivants, il y a plusieurs milliards d'années, jusqu'à aujourd'hui, les informations biologiques ont toujours été stockées sous forme d'ADN. Dans les cellules biologiques, cette information est traduite, d'abord en molécules d'ARN, puis en protéines. Jusqu'à présent, il n'existe aucun processus permettant d'inverser ce flux d'informations et de retranscrire les informations protéiques en informations ADN. Avec l'IA, cela va bientôt changer. Pour les biologistes comme moi, c'est une évolution incroyablement spectaculaire. Elle aura un impact énorme sur la biotechnologie et la médecine.