Aufnahmegerät für die Zellgeschichte
ETH-Forscher entwickeln mithilfe des Crispr/Cas-Systems einen neuartigen Speichermechanismus, der DNA-Schnipsel herstellt, die über bestimmte Zellvorgänge Auskunft geben. Der zelluläre Speicher kann künftig vielleicht sogar für Diagnosen genutzt werden.
Virenattacken, Kontakt mit Umweltgiften oder Hungerstress hinterlassen in Zellen molekulare Spuren: Solche Ereignisse verändern die Aktivität von Genen, was sich in der Boten-RNA (mRNA) widerspiegelt. In diese werden Gene übersetzt, wenn sie beim Vorgang der sogenannten Transkription aktiviert und abgelesen werden. Die Aktivität eines Gens lässt sich sehr gut anhand der in der Zelle vorliegenden mRNA-Moleküle ablesen. Allerdings verwischen diese Spuren bald: mRNA ist nicht stabil; die Zelle baut diese Moleküle oft nach kurzer Zeit ab.
DNA-Ring als Speichersystem
Forscher um Randall Platt vom Departement für Biosysteme der ETH Zürich in Basel haben nun ein molekulares Speichersystem entwickelt, das Transkriptionsereignisse speichert, und zwar in Form eines ringförmigen DNA-Moleküls, eines sogenannten Plasmids. Die gespeicherten Informationen lassen sich auslesen, in dem die Forschenden die Abfolge der DNA-Bausteine des Plasmids bestimmen.
Für ihr «Speichergerät» griffen Platts Doktoranden Florian Schmidt und Mariia Cherepkova auf das Crispr/Cas-System zurück. Dieses dient beispielsweise Bakterien und Archaeen als einfaches Immungedächtnis, das genetische Informationen über schon einmal dagewesene Krankheitserreger speichert. Das Speichern von Informationen findet an einer vordefinierten Stelle im Plasmid statt – dem sogenannten Crispr-Array.
Genetische Information wie Perlen aufreihen
In einem solchen Crispr-Array ist jeweils ein kurzes DNA-Fragment, genannt «spacer» hinterlegt, welches vom Krankheitserreger stammt. Spacer von verschiedenen Krankheitserregern sind durch kurze, identische DNA-Sequenzen, «direct repeats», voneinander getrennt, sodass das Gebilde einer Perlenkette ähnelt.
Die Forschenden haben Gene für das Crispr/Cas-System einer anderen Bakterienart in das Erbgut des Darmbakteriums Escherichia coli eingebaut. Eines dieser Cas-Gene ist mit einer Reversen Transkriptase verbunden, einem Enzym, welches nach der Vorlage von RNA-Molekülen DNA-Moleküle mit demselben Informationsgehalt herstellt, das heisst RNA-Information in DNA-Information zurückübersetzt.
Mit diesen Fremdgenen ergänzte Escherichia coli-Zellen waren in der Lage, einen molekularen Proteinkomplex herzustellen, der kurze mRNA-Moleküle bindet. Die Reverse Transkriptase übersetzt solche kurzen RNA-Moleküle in DNA-Fragmente, welche die selbe Information wie die originale RNA tragen und anschliessend analog zum natürlichen Prozess in das CRISPR-Array eingebaut werden. Dieser Prozess kann mehrfach stattfinden und neue Spacer werden in das Crispr-Array in absteigender chronologischer Reihenfolge eingebaut: Das zeitlich zuletzt aufgenommene DNA-Fragment sitzt stets zuvorderst.
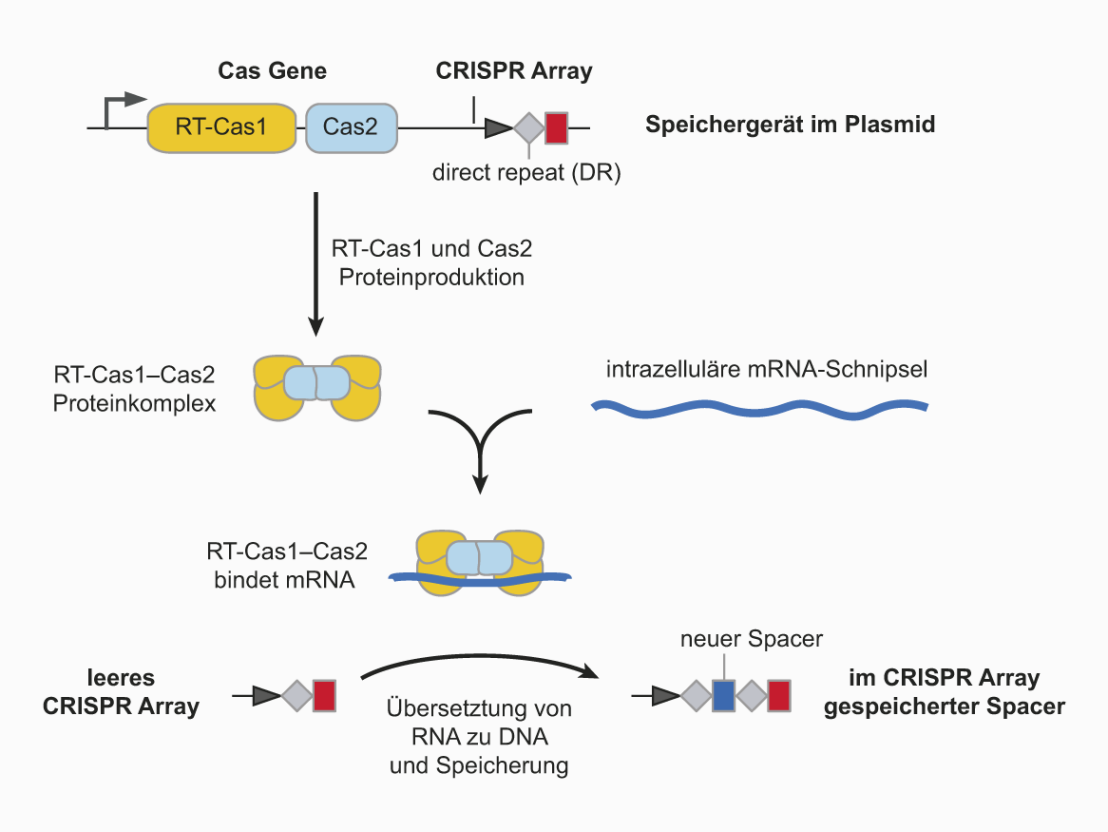
Auf diese Weise lassen sich an sich beliebig viele DNA-Fragmente speichern. Da DNA sehr stabil ist, wird die darin gespeicherte Information für lange Zeit aufbewahrt und zudem von einer Bakteriengeneration an die nächste weitervererbt. Durch eine Bestimmung der DNA-Bausteine in den erzeugten Plasmiden können die Forschenden schliesslich analysieren, welches Gen wie oft und in welcher zeitlicher Abfolge aktiv war.
«Dieses System ist ein biochemischer Datenlogger. Damit können wir über mehrere Generationen die Genantwort der Bakterien auf äussere Einflüsse aufzeichnen und abrufen», sagt Florian Schmidt, Erstautor der entsprechenden Studie, die soeben in der Fachzeitschrift «Nature» erschienen ist. «Schon lange haben weltweit Forscherinnen und Forscher an solchen künstlichen zellulären Datenloggern gearbeitet. Wir sind aber wohl die ersten, die nun einen entwickelt haben, der eine solche Vielfalt von Signalen aufnehmen kann», sagt ETH-Professor Platt nicht ohne Stolz. Die Basler Forschenden haben über zwei Jahre an diesem System gearbeitet.
Ganzes Logbuch auswerten
Bisher mussten sich Forschende oft mit einer Momentaufnahme aus dem Leben einer Zelle begnügen. Um dieses Bild aufzunehmen, müssen sie in der Regel die Zellen zerstören, mRNA extrahieren und diese aufwändig quantifizieren. Im Crispr-Array zeichnen die Zellen jedoch einen Verlauf über mehrere unterschiedliche Stadien auf, sodass die Forscher quasi ein ganzes Logbuch auswerten können.
Im Rahmen ihrer Studie zeigten die ETH-Forschenden, wie mit dem Datenlogger ausgestattete E.-coli-Bakterien auf das Herbizid Paraquat reagieren. Das Gift löste in den Zellen eine Genantwort aus, welche die Wissenschaftler auch noch Tage später aus dem Crispr-Array auslesen konnten. Ohne Datenlogger wären die molekularen Spuren, die auf den Kontakt mit dem Herbizid hinweisen, dann längst abgebaut gewesen und die Information damit verloren.
Zur Anwendung kommen könnte der biochemische Logger in erster Linie in der Forschung, es ist aber auch denkbar, dass er als eine Art Sensor zur Messung von Umweltgiften, wie beim Beispiel des Herbizids, oder in der Diagnostik zum Einsatz kommt. Der Weg zu einer praktischen Anwendung ist allerdings noch weit. Die vorliegende Studie ist erst ein Machbarkeitsbeweis. Randall Platts Forscher sind aber derzeit daran, das System auf andere Zelltypen zu übertragen, um den Weg hin zu diagnostisch nutzbaren Werkzeugen zu ebnen.
Literaturhinweis
Schmidt F, Cherepkova MY, Platt RJ. Transcriptional recording by CRISPR spacer acquisition from RNA. Nature, 3. Oktober 2018, doi: externe Seite 10.1038/s41586-018-0569-1
Kommentare
Noch keine Kommentare